
无刺蜂中发现线粒体基因组结构新类型
真核生物类群线粒体拥有独立的遗传物质,线粒体基因组结构在真核生物中表现出极高的多样化,其中动物的线粒体基因组是保守的双链环状DNA分子,基因组大小为15~18kb,基因含量包括13个蛋白编码基因(protein coding genes,PCGs)、2个核糖体rRNA编码基因(ribosomal RNA, rRNAs)和22个转运tRNA编码基因(transfer RNA genes, tRNAs)。随着越来越多的动物线粒体基因组被解码,研究人员发现不少的类群中存在多样化的结构变异和基因重排(通常是tRNA)。为进一步了解动物线粒体基因组的结构变异和演化模式,及其对系统发育重建的影响,中国科学院西双版纳热带植物园(以下简称“版纳植物园”)的研究团队选取3个社会性蜂类类群,无刺蜂(麦蜂族,Meliponini)、熊蜂(熊蜂族,Bombini)和蜜蜂(蜜蜂族,Apini),作为研究对象,基于系统发育学框架,进行线粒体基因组的比较分析,旨在揭示其结构变异和演化历史。
系统发育分析表明,3个社会性蜂类最早分化的是蜜蜂族,随后麦蜂族和熊蜂族为姐妹关系。麦蜂族系统发育树可以进一步分为印度-马来/澳大利亚区分支、热带非洲区分支和热带美洲区分支。蜜蜂和熊蜂的线粒体基因组均为动物类群的典型结构,而无刺蜂类群表现出较大的结构变异,其印度-马来/澳大利亚区分支所有物种的线粒体基因组具有反向重复(inverted repeats, IR)区,并且存在大量PCGs和rRNAs基因重排,研究团队通过设计特定引物扩增IR区和单拷贝区(SC)交界处的序列,证明了IR区存在的真实性(图1)。在热带美洲区分支中,包括物种Frieseomelitta varia、Tetragonisca angustula和Nannotrigona testaceicornis的一个单系表现出与印度-马来/澳大利亚区分支不同式样的PCGs和rRNAs基因重排(图2)。祖先状态重建分析表明,线粒体基因组结构变异均为一次性地发生在两个分支各自的最近共同祖先节点(图2)。IR区的出现使得PCGs和rRNAs基因重排在印度-马来/澳大利亚区分支展示出了多样性,而常见tRNA重排在3个类群中都被发现,并存在重排热点区。与以往研究以单一物种代表属及以上水平的分类阶元的研究方法不同,该研究对物种和种群水平进行了更为密集的采样(74种的154个样本),发现tRNA重排存在显著的种间变异(图3),表明更全面的采样对更深入理解线粒体基因组结构变异和基因重排模式,以及利用其进行系统发育重建的重要性。
该研究成果以“Comparative analyses of mitogenomes in the social bees withinsights into evolution of long inverted repeats in the Meliponini(社会性蜂类的线粒体基因组比较分析以及麦蜂族中反向重复区结构的演化)”为题发表在期刊Zoological Research《动物学研究》上。论文第一作者李虞冉(现为协同进化研究组在读博士研究生,指导老师为彭艳琼研究员)在硕士研究生期间完成的相关研究工作,指导老师Richard Corlett研究员和郁文彬研究员为共同通讯作者,合作导师汪正威研究员为第二作者。该研究得到了中国科学院战略性先导科技专项,中国科技部基础调查专项,云南省“兴滇英才支持计划”和版纳植物园“十四五”科技创新规划的支持。
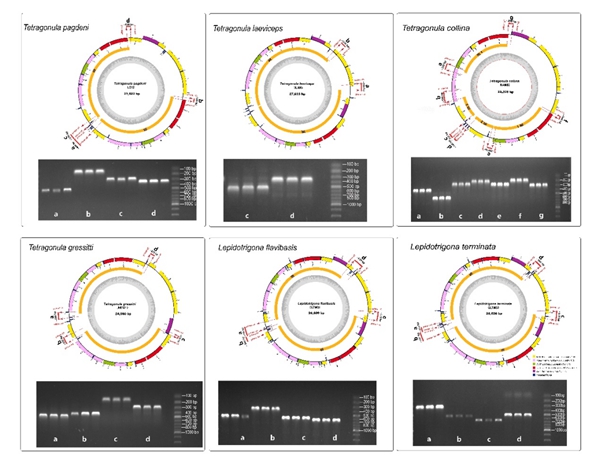
图1. 针对6种无刺蜂进行IR区验证设计的引物分布情况,及产物的凝胶电泳图
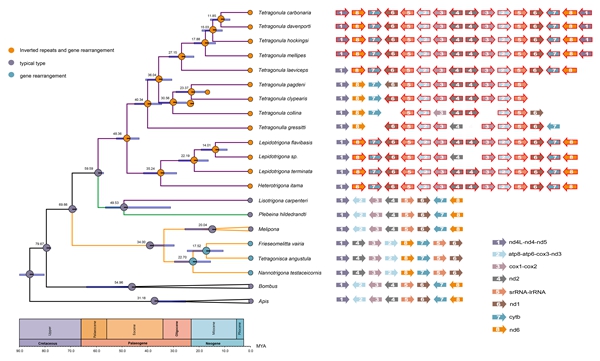
图2. 三个社会性蜂类线粒体基因组结构变异的祖先状态重建

图3. 三个社会性蜂类线粒体基因组的基因重排模式图
Copyright 2002-2022 中国科学院西双版纳热带植物园【滇ICP备13004273号-1】



